 京都大学は,天然に存在する膜蛋白質に蛍光色素等の目印を付けるための分子ツールを開発した(ニュースリリース)。特に,実際に生きた細胞の膜表層に存在する蛋白質に,遺伝子操作などの余分は処理を施す事なく目印をつけることで,極めて天然に近い環境下で,蛋白質のイメージング(可視化)や,寿命,分解過程を解析することに成功した。
京都大学は,天然に存在する膜蛋白質に蛍光色素等の目印を付けるための分子ツールを開発した(ニュースリリース)。特に,実際に生きた細胞の膜表層に存在する蛋白質に,遺伝子操作などの余分は処理を施す事なく目印をつけることで,極めて天然に近い環境下で,蛋白質のイメージング(可視化)や,寿命,分解過程を解析することに成功した。
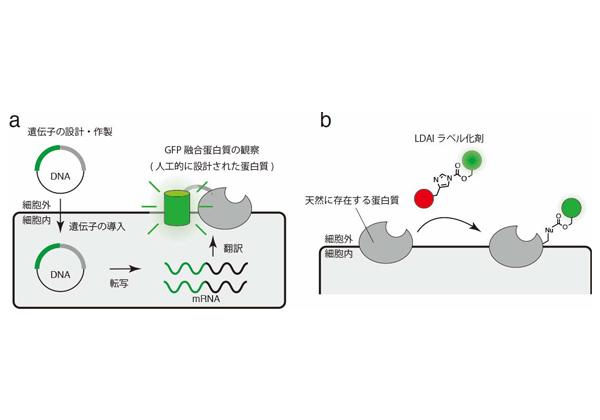
これまで,細胞に存在する蛋白質を可視化し観察するための手法として,GFP(緑色蛍光蛋白質)を使った遺伝子工学的な手法を用いる必要があった。この手法は,人為的に設計し作製された遺伝子を細胞内に導入し,観察したい蛋白質に似た『モデル蛋白質』を観察するもの。従って,もともと細胞に存在している蛋白質をそのまま観察することは不可能で,本来の蛋白質の挙動を観察できる技術が望まれていた。
これまで研究グループは,天然に存在している蛋白質そのものを,遺伝子工学を用いずに有機化学によって蛍光色素等で目印をつける『LDAIラベル化法』の開発を行なってきた。これは,ラベル化剤と呼ばれる分子を細胞にかけるだけで,特定の狙った蛋白質のみに目印を付けて可視化を可能とし,解析する手法。
今回,LDAIラベル化法がガンやその他の疾病に深く関連するため創薬の標的として注目されている,様々な膜蛋白質に広く適用でき,天然に存在する膜蛋白質を可視化(イメージング)し動態を観察できるだけでなく,それぞれの膜蛋白質の寿命やその分解過程を解析することのできるツールであることを実証することに成功した。
従来の手法に比べて簡便で,極めてナチュラルな条件で蛋白質の解析ができるため,膜蛋白質の細胞内での挙動や分解と深く関わる疾病のメカニズムの解明に役立つと期待される。
関連記事「日本板硝子,少量サンプルの高感度測定が可能な蛍光検査器を開発」「NIMSと慶大,生命活動や病気に広く関わる補酵素の蛍光イメージングに世界で初めて成功」